データ駆動型生物学 (作村研究室)
-
研究室ホームページ
- http://ncb.naist.jp/
-
全学オンラインセミナー
2022年度に開催されたBio Discovery Session(全学オンラインセミナー)のアーカイブ動画の一覧です。 - Bio Discovery Session
-
Webメディア
NAIST Edge BIOは、奈良先端科学技術大学院大学 バイオサイエンス領域 の各研究室で取り組んでいる「最先端」の研究プロジェクトや研究成果について、研究者だけではなく受験生や一般の方にも分かりやすく紹介するためのWebメディアです。 - NAIST Edge BIO 第21回
研究・教育の概要
本研究室では、実験観測データを数理的に解析し、分子レベルから人の集団レベルに至る現象の背後にある法則を解明することを目的としています。情報・数理解析を活用することで、異なるスケールの現象を定量的に関連付け、生命機能をさまざまな物理量間の相互作用として理解することを目指します。これらの研究を通じて、データ解析の手法を習得するとともに、自主的な思考力を養っていきます。
主な研究テーマ
細胞の変形と移動のシステムバイオロジー(分子と細胞)
神経細胞が生み出す活動電位は、膜タンパク質と膜電位からなるシステムの一側面です。同様に、細胞の変形や移動も、分子・力・形態が相互作用するシステムと捉えられます(図1)。これらの異なる物理単位のシグナルを解明するには、データ間の数理的関係を導き出すことが重要です。本研究室では、以下のテーマに取り組んでいます。
- 細胞骨格形成および力を制御する分子と細胞変形のシステム解明
- 機械学習を用いた細胞の牽引力推定
- 細胞の弾塑性体特性の定量的モデリング
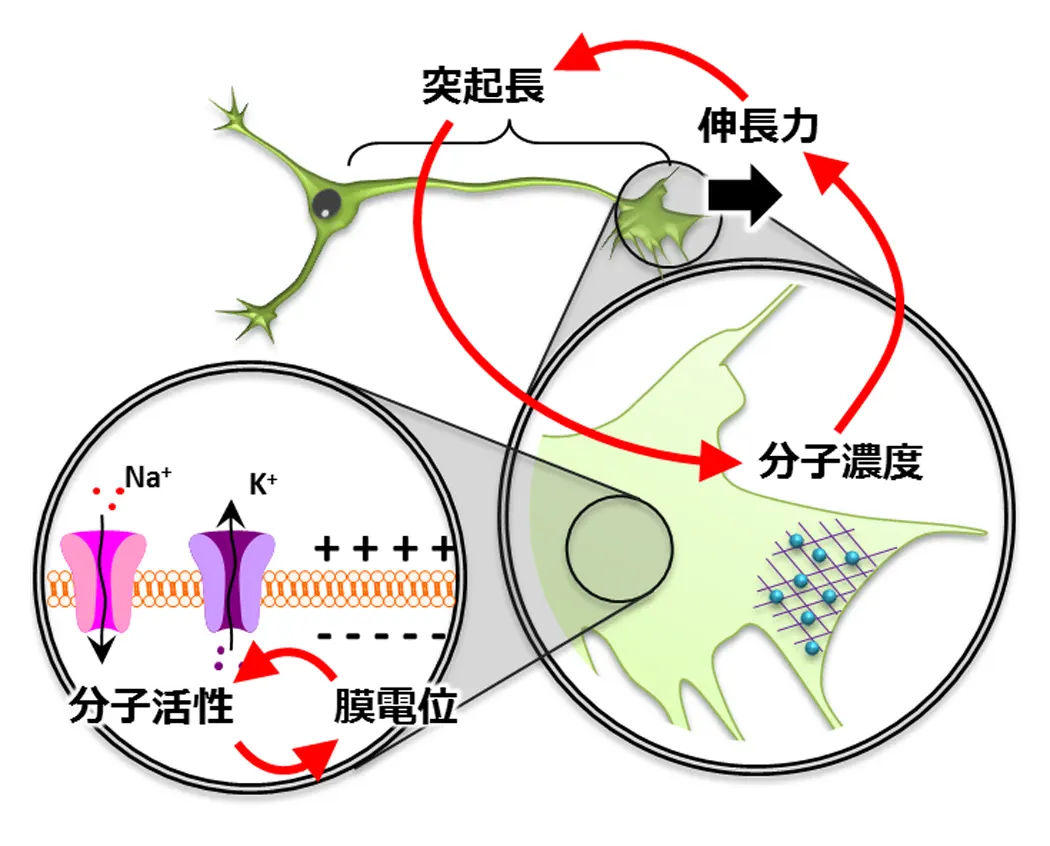
(図1) 膜電位・分子からなるシステム、神経突起長・力・分子からなるシステムの例。様々な物理量(表現型)へのシグナル変換は、実験データから導出される。これらのシグナル変換を組み合わせればシステムが再構成される。
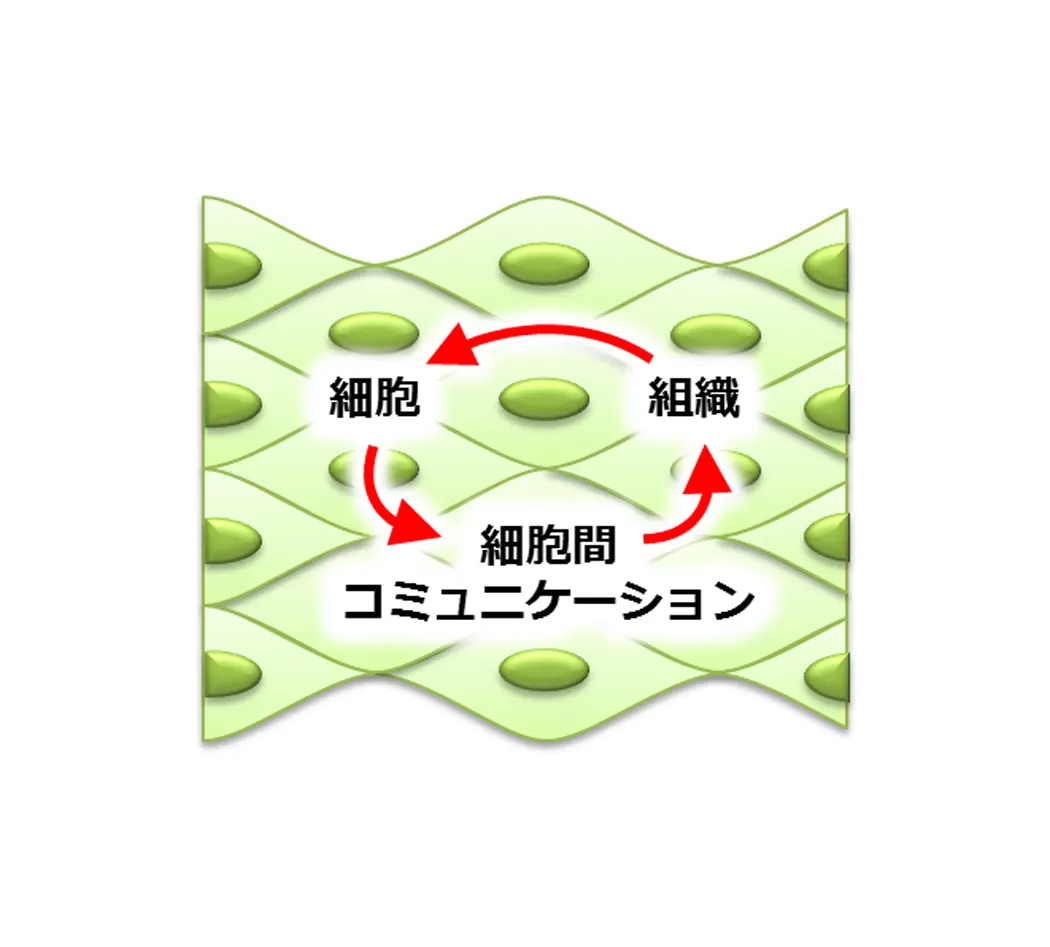
生体組織形成のシステムバイオロジー(細胞と組織)
生体組織は、細胞と組織が一体となって機能するシステムです(図2)。実験データを用いた数理解析により、細胞社会がロバストではなくレジリエンスな組織機能を生み出す原理の解明を目指します。本研究室では、以下のテーマに取り組んでいます。
- 脊椎動物の発達における細胞間協調
- 細胞移動の原理に基づく血管新生
医療システムバイオロジー(分子・組織から人の疾患)
医療データを数理解析し、分子・組織レベルから人の疾患に至るシステムの解明を目指します。呼気や血液成分、組織画像を活用した疾患診断・予測に加え、機械学習を用いた解析を行います。また、大規模医療レセプトデータを用いて、疾患要因の推定や疾患間の関連性の抽出を行います。以下のテーマに取り組んでいます。
- 呼気・血液成分を用いた疾患診断・予測
- 大規模医療データと機械学習による疾患リスクの算出
- MRI 画像を用いた疾患推定
- 疾患関連遺伝子の抽出
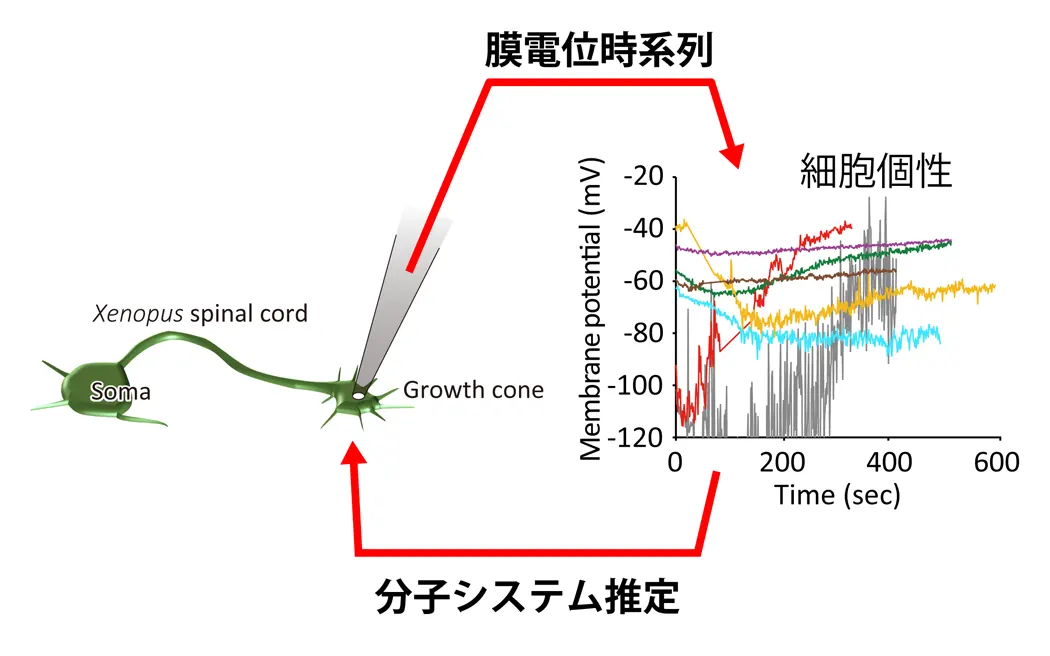
(図3) 膜電位時系列から細胞内分子システムの推定。細胞内の分子間相互作用を調べる実験は非常に困難であるが、膜電位計測は比較的容易である。膜電位時系列を用い、計算機により細胞内分子システムを推定する。
生物データ解析のための前処理と機械学習応用
複雑なデータから細胞や個体に共通する知見を抽出することは、もはや研究者の直感だけでは困難です。本研究室では、データ前処理技術や機械学習を活用し、データに潜む法則や現象を引き起こす因子の推定など、以下のテーマに取り組んでいます。
- トランスオミクス解析
- 細胞が生む機械的力の機械学習による推定
- 組織形成に関わる遺伝子制御ネットワーク推定
- モデル生物のデータ駆動型繁殖制御
主な発表論文・著作
- Suzuki et al, Sci Rep, doi:10.1038/s41598-025-91034-8, 2025
- Ohkubo et al, Sci Rep, doi: 10.1038/s41598-024-76029-1, 2024
- Fujikawa et al, Biophys J, doi: 10.1016/j.bpj.2023.10.032, 2023
- Ohkubo et al, Sci Rep, doi: 10.1038/s41598-023-40469-y, 2023
- Itoh et al, Sens Actuators B Chem, doi: 10.1016/j.snb.2023.133803, 2023
- Kunida et al, Cell Rep, doi: 10.1016/j.celrep.2023.112071, 2023
- Minegishi et al, J Vis Exp, doi: 10.3791/63227, 2021
- Wong et al, Plant Physiol, doi:10.1104/pp.19.00811, 2019
(こちらのリンクはすべて外部サイトへ遷移します![]() )
)
 奈良先端科学技術大学院大学
奈良先端科学技術大学院大学